| Département : Biologie, Signaux et Systèmes en Cancérologie et Neurosciences Responsable scientifique : Laurent KOESLER Thèses associées 2016–2021 : Angélique VOLFART (2020) ; Samuel LOUVIOT, Luna ANGELLI, Vivinia DEL ROCCIO (en cours) |
Contexte
Actuellement la prise en charge des pathologies cérébrales reposent sur la confrontation de données médicales diverses et multiples. En effet, afin de proposer un diagnostic fiable et précis, les médecins doivent mettre en correspondance et interpréter des données d’imagerie anatomique (IRM, Scanner, . . . ), fonctionnelle (EEG, IRMf, ...) et métabolique (PET, TEMP, . . .) en plus des données cliniques rapportées par le patient. Le référentiel, les dimensions, le format des données sont très souvent variables d’un système d’imagerie à l’autre ce qui rend très difficile en clinique la fusion de ces images dans un même espace anatomique propre au patient. Cette problématique est un enjeu technologique majeur pour le diagnostic des pathologies cérébrales car de cette fusion multimodale aboutira une hypothèse concordante ou discordante et donc à un traitement spécifique associé.
Objectifs scientifiques
Les objectifs scientifiques et méthodologiques sont de recaler, représenter, localiser et caractériser certaines structures cérébrales et / ou électrodes grâce à différentes modalités d’imagerie (anatomique et fonctionnelle). Pour cela, nous avons développé une plateforme logicielle qui répond à cette problématique. Il s’agit d’ICEM pour Imagerie Cérébrale Multimodale qui s’inscrit dans l’aide au diagnostic médical, à la planification préopératoire et à la décision chirurgicale.
Description
Le logiciel effectue un recalage 3D de plusieurs modalités d’imagerie et autorise la visualisation par transparence, la navigation 3D et l’exploration de données. Les modalités d’imagerie actuelles prisent en compte sont :
— IRM anatomique et fonctionnelle (IRM(f)) ;
— scanner (CT) ;
— imagerie de sources électriques (ISE) ;
— stéréo-encéphalographie (SEEG);
— tomographie par émission de positons (TEP).
Du point de vue du traitement du signal, des fonctionnalités ont été intégrées dans deux domaines qui sont la détection automatique et l’analyse statistique. Elle permet :
— fusion de l’IRM préopératoire et du CT post-opératoire ;
— détection et Localisation automatique des électrodes (multi-capteurs) intra-cérébrales (SEEG);
— recalage des électrodes de scalp (EEG);
— labélisation des électrodes et identification anatomique des structures cérébrales explorées ;
— export des données dans un repère anatomique commun standardisé (MNI ou Talairach) ;
— traitement statistique automatique de type Signal Processing Mapping SPM;
— test ANCOVA comparant les données TEP du patient à une base de données spécifique (CHU Nancy) de sujets sains ;
— affichage et caractérisation des zones d’hypo- et d’hyper-métabolismes statistiquement pertinentes.
Ces développements sont intégrés à une forge logicielle partagée.
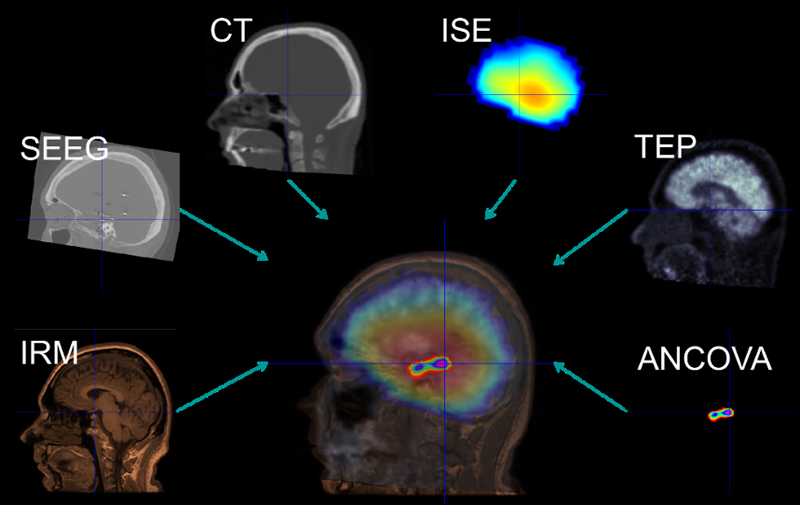 |
| FIGURE 81 – Les différentes modalités prises en compte dans le logiciel |